7月31日,中国科学院深圳先进技术研究院合成生物学研究所胡政课题组与厦门大学数学科学学院周达课题组合作,在国际学术期刊Nature Biotechnology(自然生物技术)发表了题为PhyloVelo enhances transcriptomic velocity field mapping using monotonically expressed genes的研究论文,提出一项新颖的单细胞分化轨迹推断的算法框架,命名为PhyloVelo。该方法通过联合单细胞转录组数据和谱系示踪数据,鉴定随细胞分裂而表达量呈单调递增或递减的基因,即单调表达基因,并利用演化方法估计单调基因的RNA转录变化速度,以构建单细胞转录组速度场(RNA velocity fields),实现对细胞分化轨迹的高精准推断。
细胞分化和命运决定是生命的奥秘之一,也是生命科学领域的核心问题,揭示其规律和机制对于理解生命现象和促进医学发展具有重要意义。然而,准确地追踪细胞的动态分化轨迹并非易事,尤其是在疾病等干扰情况下的细胞命运转换过程,存在较大的随机性和难以预测性。当前,单细胞转录组测序(scRNA-seq)是研究细胞分化的一种强有力的技术,可以通过分析单个细胞的转录组状态来推断细胞分化的动态过程。尤其是基于信使RNA(mRNA)的剪切动力学而发展起来的RNA速度模型(RNA velocity),可预测单细胞转录组在“过去”或“未来”时间的状态,是单细胞轨迹推断的经典算法。然而由于单细胞测序技术与mRNA转录/剪切动力学的高度复杂性,RNA速度模型在实际应用中常常出现错误推断或不稳健的问题。
在本文中,作者提出利用单细胞系统发育树的枝长信息代替物理时间,联合单细胞转录组数据,可以研究基因表达随着枝长(即时间)的变化规律,特别是鉴定随细胞分裂而表达量呈单调递增或递减的基因(即单调表达基因),其RNA变化速度可以利用分子演化模型进行定量估计,从而构建单细胞转录组速度场,实现对细胞分化轨迹的高精准推断(图1)。
PhyloVelo算法主要分为两个部分。第一步是单调表达基因(monotonically expressed gene, MEGs)的识别。利用了单细胞转录组数据和细胞系统发育树(cell phylogeny)信息,通过带漂移的扩散过程模型(diffusion process)和零膨胀的负二项分布(ZINB)来估计每个基因在系统发育树上各个时间点的表达水平。然后,利用表达水平估计值和对应系统发育树时间的相关性,筛选出那些随着谱系时间线性单调增加或减少的基因,即单调表达基因MEGs(图1)。第二步是基因表达变化速度的估计。本算法假设每个MEG的表达水平随着时间的变化速率是恒定的,即扩散方程中的漂移系数是常数。通过最大似然估计,可以得到每个MEG的漂移系数,从而得到每个细胞的单调基因表达变化速度的向量。将这个向量映射到降维空间(tSNE, UMAP等)重建RNA速度场,可推断每个细胞在过去一个单位时间(例如一个细胞分裂或突变)的转录组状态,即逆时间重建细胞分化轨迹(图1)。
作者在多种仿真数据和真实数据上对PhyloVelo算法的准确性和鲁棒性进行了验证,PhyloVelo都能够高准确地推断仿真数据中的线性、分叉和趋同等复杂分化结构,与真实分化轨迹高度一致(图2)。此外,PhyloVelo还在小鼠早期胚胎发育中表现出优于RNA velocity的性能,能够准确地识别出红细胞系列中血液/内皮祖细胞作为最早期的细胞类型,并且与细胞增殖能力呈强相关(图3)。
除了小鼠胚胎发育外,PhyloVelo还准确解析了小鼠和人类的肿瘤演化、免疫细胞动态发育等其他生物过程中的复杂分化轨迹,并量化了细胞类型之间的状态转换概率。例如,在肺癌模型中,PhyloVelo揭示了癌细胞的逆向分化轨迹。在抗PD-1治疗后的CD8+ T细胞中,PhyloVelo发现活化CD8+ T细胞的来源在免疫治疗前后有明显的变化,表明T细胞高度的命运可塑性。
总之,PhyloVelo是一种利用单细胞谱系和转录组数据重建细胞命运转变的新方法,具有高度的准确性和鲁棒性。PhyloVelo不仅可以克服传统RNA velocity方法的局限性,还可以发现隐藏在转录组数据中的“时钟基因”,为揭示细胞分化的分子机制提供线索。PhyloVelo为研究生物发育和疾病进展提供了一个有力的工具,也为未来的单细胞谱系和转录组数据分析提供了一个新的视角。为了方便读者使用PhyloVelo方法,研究团队还在线发布了使用手册和具体应用案例,详见网站: https://phylovelo.readthedocs.io/en/latest/ 。
中国科学院深圳先进技术研究院合成生物学研究所胡政研究员与厦门大学数学科学学院周达副教授是该论文的共同通讯作者,中国科学院深圳先进院和厦门大学联培博士生王琨是论文的第一作者。中山大学贺雄雷教授、中国科学院动物研究所翟巍巍研究员、美国斯坦福大学Christina Curtis教授、以及深圳先进院合成生物学研究所资治科研究员对本研究提供了宝贵的指导和意见。本项研究获得科技部国家重点研发计划、国家自然科学基金、广东省杰出青年基金、中央高校基本科研专项资金、中国博士后基金及深圳合成生物学创新研究院等项目的支持。
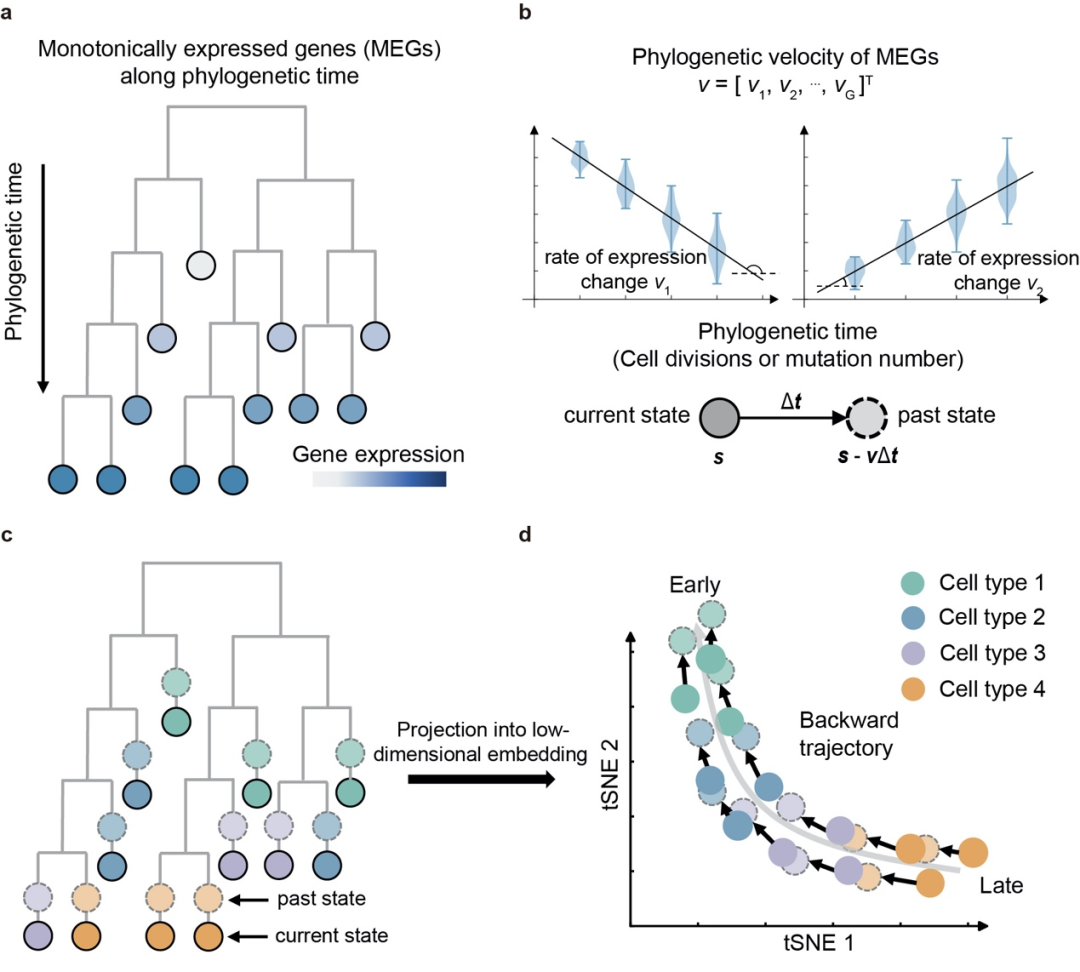
图1 | PhyloVelo算法框架示意图
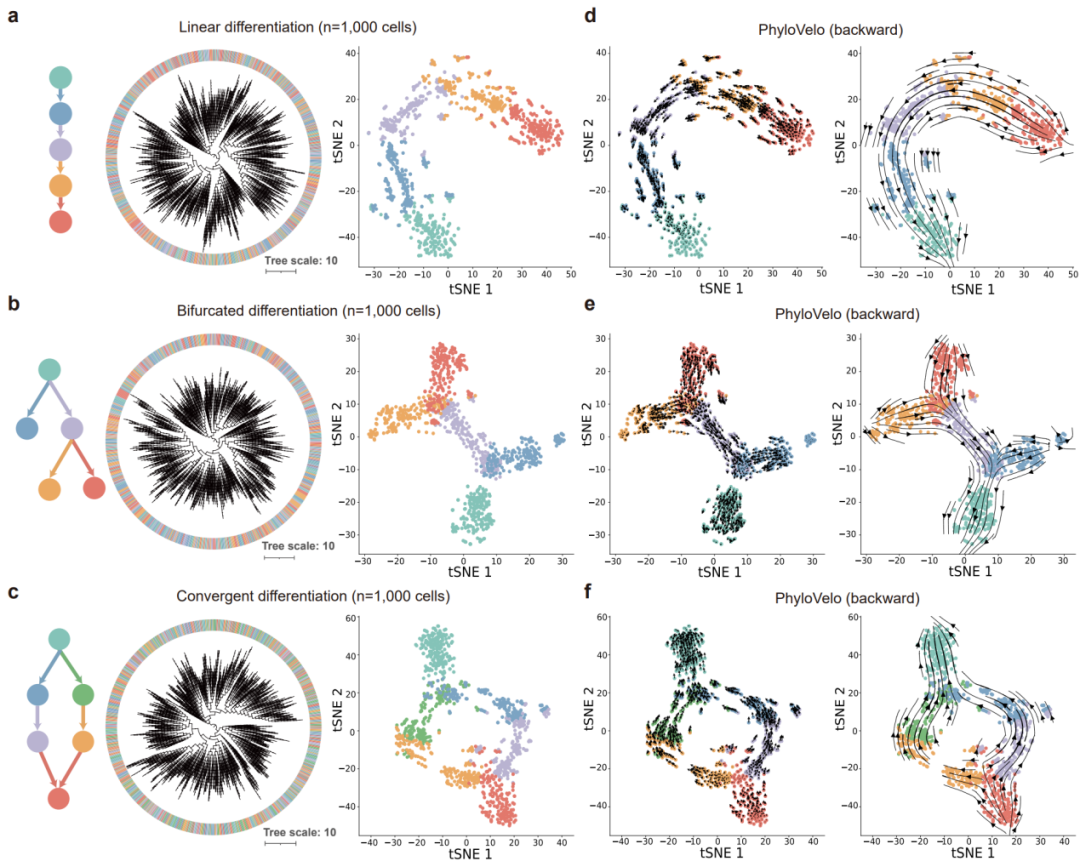
图2 | PhyloVelo准确推断仿真数据中的细胞分化轨迹
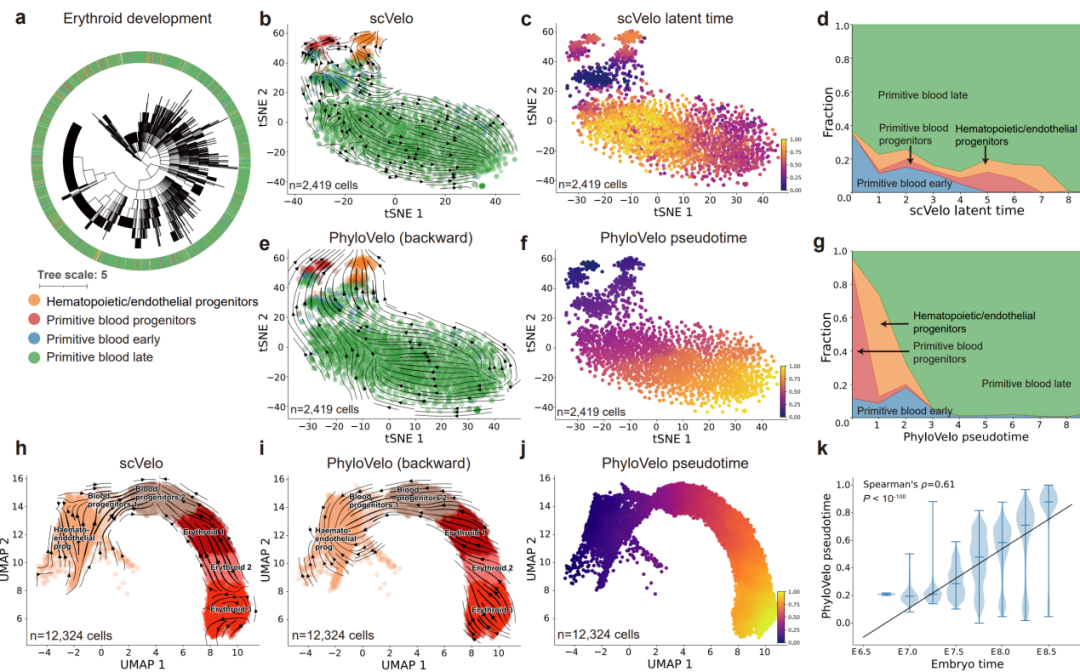
图3 | PhyloVelo重构小鼠血液系统发育的细胞分化轨迹
图4 | PhyloVelo使用手册网站截图
附件下载:


